En torno a los datos
Presentación del capítulo
En este capítulo vamos a recorrer varias técnicas y representaciones de datos estructurados (tablas de clases y números) de manera complementaria a todo lo que ya hemos visto.
Con respecto a la interpretación de datos, el punto de vista que hemos abordado ha consistido en preparar los datos para su uso en un algoritmo supervisado o no supervisado, en gran parte para hacerlos inmanejables por ese algoritmo. A petición de nuestros lectores, vamos a interesarnos por la manipulación de los datos en sí, tanto para elaborarlos como para poder realizar hipótesis iniciales o tener unas primeras intuiciones sobre su significado. Obviamente, los dos puntos de vista son complementarios.
El diseño de este capítulo no se debe interpretar como un «protocolo» o método de análisis de datos. Hemos hecho una elección didáctica que introduce paso a paso diferentes herramientas o que presenta lo mismo desde diferentes ángulos, a fin de que el lector tenga elementos adi-cionales a lo que ya conoce para que se sienta a gusto, confiado e imagine su propio sistema exploratorio, según las circunstancias.
Captura directa y rápida de un dataset
Para evitar redundancias innecesarias, vamos a equiparnos con un dataset cuyos datos se utilizarán en el capítulo, que trata sobre la creación de un modelo sin programación usando una herramienta como BigML. El paquete mlbench permite acceder a estos datos y también construir datasets adaptados a un tipo de problema.
library(tidyverse)
library(mlbench) # paquete para crear o acceder a los datos de prueba Puede enumerar fácilmente todos los datasets de demostración que se ofrecen en los paquetes instalados en una máquina.
data(package = .packages(all.available = TRUE)) # lista de los datasets disponibles A continuación, se muestra un (muy) breve extracto de lo que está disponible en nuestra máquina:
...
Data sets in package ‘mlbench':
BostonHousing Boston Housing Data
BreastCancer Wisconsin Breast Cancer Database
DNA Primate splice-junction gene sequences (DNA)
Glass Glass Identification Database
HouseVotes84 United States Congressional Voting Records 1984
Ionosphere Johns Hopkins University Ionosphere database
LetterRecognition Letter Image Recognition Data
Ozone Los Angeles ozone pollution data, 1976
PimaIndiansDiabetes Pima Indians Diabetes Database
... Vamos a invocar los siguientes datos:
data(PimaIndiansDiabetes) # acceso a los datos de diabetes de los indios
df <- data.frame(PimaIndiansDiabetes) Contiene información sobre la diabetes de las mujeres de una tribu india de América del Norte (los Pimas...
Análisis de la conformación de las distribuciones respecto a la distribución normal
Podemos validar algunas de nuestras intuiciones respecto a la normalidad de los datos comparando estas densidades con la ley o distribución normal mediante un diagrama Cuantil-Cuantil.
# ajustes de la ley normal (mu,e)
plot_qq(df) # diagramas Cuantil-Cuantil 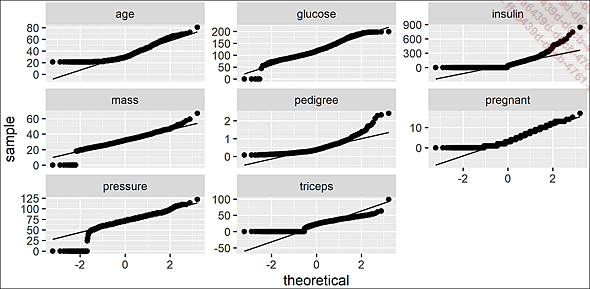
Diagramas Cuantil-Cuantil
Incluso sin saber cómo interpretar con precisión un diagrama de este tipo, llama nuestra atención sobre ciertas características de los datos:
-
Habrá que analizar los valores nulos (¿cero o desconocido?) dentro de glucose, insulin, mass, pregnant, pressure, triceps, que nos permitirán decidir nuestra estrategia (sustituir por NA, por una media, eliminar filas de mala calidad, hacer un tratamiento particular de los outliers, etc.).
-
El hecho de que glucose, mass y pressure parezcan líneas rectas cercanas a la diagonal (excluyendo los valores nulos que se deben cualificar) puede hacer pensar que, de acuerdo con su apariencia general en la figura Densidades por variable, son más o menos cercanas a una ley normal.
-
La forma regular de pedigree podría alentarnos a relacionarla con otra ley (que se debe probar), normalmente una ley de Poisson o Lognormal.
Este ajuste se puede comparar distinguiendo entre los casos «con» o «sin» diabetes.
# comparando...Dependencia lineal entre variables
De forma adicional, es habitual comprobar si algunas de las variables explicativas no están fuertemente correlacionadas, lo que podría provocar que se prueben modelos de predicción que mantendrían solo una u otra de estas variables (para hacerlo bien, comparamos la eficiencia con ambas y, cuando es similar, nos quedamos con la variable que es más fácil de interpretar o recopilar en el mundo real).
plot_correlation(df) # una tabla de correlaciones 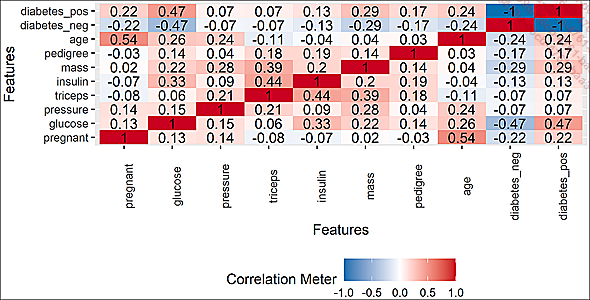
Correlación dos a dos
Nuestro problema no es linealmente trivial; de forma clara, la diabetes no se correlacionada solo con una variable. Exceptuando la variable age, parece completamente descartado que exista alguna relación más o menos lineal y simple entre el número de embarazos y el resto de las variables explicativas. Podemos representar esto, quizás erróneamente, imaginado dos grandes familias de predictores: «sociales» (edad, número de embarazos) y técnicos (el resto). Dicho todo esto, tenga en cuenta que esta pista solo se ocupa de las relaciones lineales, que no es el caso más común en la realidad. Recordemos también que la calidad de los predictores no nos permite sacar conclusiones sobre la noción de causalidad.
Ahora bien: nada nos impide formarnos una opinión personal sobre la monotonía de la relación entre dos variables...
Resalte de las diferencias entre las distribuciones
Ahora intentaremos resaltar (o no) las posibles diferencias en las distribuciones para ciertas variables de aspecto gaussiano (para este ejemplo, hemos elegido glucose) entre la distribución con diabetes o sin ella.
Nuestra principal preocupación será la importancia de cualquier diferencia en la media. La herramienta también nos aporta información sobre la diferencia en la desviación típica, que no requiere una interpretación fuerte, sino que el hecho de que sea significativa puede reforzar la idea de que un algoritmo bien elegido podrá mejorar la predictibilidad de diabetes
library(lessR)
d <- df # por defecto lessR utiliza d como dataset
tt.brief(glucose ~ diabetes) # gráfico y estados 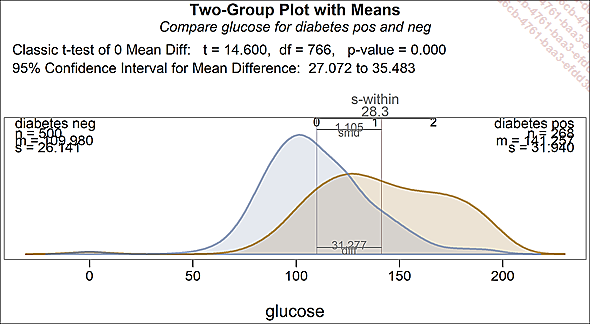
Distribuciones de la glucosa en función de la diabetes
La diferencia en la media es significativa
También es posible que desee una representación de «bigote/violín», utilizando la siguiente sintaxis:
library(ggstatsplot)
set.seed(666)
ggbetweenstats(
data = df,
x = diabetes,
y = glucose,
title = "Distribución de la glucosa con o sin diabetes"
) 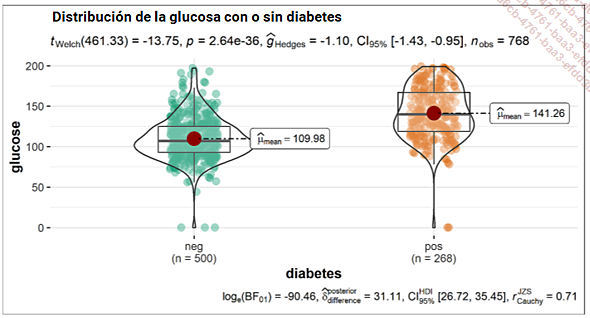
Diagrama de bigotes/violín comparado
También podemos...
Puntos atípicos
Ya hemos trabajado en la identificación de valores atípicos u outliers. La siguiente representación permitirá extraer observaciones candidatas para calificarlas como puntos atípicos. Mediante pruebas sucesivas de diferentes parámetros, puede evaluar el mayor o menor poder de discriminación del contorno que va a dibujar. Falta por estudiar de manera individualizada los puntos exteriores, cuyo identificador está especificado, para comenzar a vislumbrar su estrategia de tratamiento de este tipo de puntos.
Mantener puntos técnicamente atípicos u extraños (ya sean anomalías o no) se puede considerar válido o no, dependiendo del tratamiento que se aplique a estos valores. Mantener o no los puntos correspondientes a observaciones excepcionales puede ayudarlo a reducir el overfitting o, por el contrario, disminuir la predictibilidad de sus modelos. Cualquier decisión de este tipo se debe registrar cuidadosamente como una hipótesis estructurante en la documentación de sus modelos. A menudo tendrá que verificar estas suposiciones y cambiar su posición sobre ellas para mejorar su trabajo. No tome al pie de la letra las afirmaciones que pueda encontrar por ahí, aunque parezca que le proporcionan un método automático para manejar los valores outliers (lo que no le impide crear un método y automatismos...
Ordenaciones y agregaciones
1. Ordenaciones automatizadas
Ahora vamos a estudiar diferentes ordenaciones (incluida la ordenación plana) de nuestros datos a través de varias funciones especializadas.
Para ayudarnos en la presentación de nuestros resultados, hemos escrito una función naive de presentación, que vamos a utilizar varias veces en esta sección:
# una pequeña función para imprimir head + tail de un objeto dato
h_t <- function(x, titulo = "", sub_titulo ="", n = 3)
{
cat(titulo, "\n")
cat(sub_titulo, "\n")
print(rbind(head(x,n),tail(x,n)))
} Proponemos visualizar la distribución de elementos entre el número de embarazos (que fue el resultado de un conteo, y por lo tanto una variable cuantitativa ordenada) y la diabetes. La ordenación más sencilla se realiza usando la instrucción R table de la siguiente manera:
# ordenación plana sobre 2 var discretas
t <- table(df$pregnant,df$diabetes) # ordenación sencilla
h_t(t,
"6 líneas extraídas de una simple ordenación plana, núm embarazos
vs estado respecto a la diabetes",
"----------"
)
6 líneas extraídas de una simple ordenación plana, núm embarazos vs
estado respecto a la diabetes
----------
neg pos
0 73 38
1 106 29
2 84 19
14 0 2
15 0 1
17 0 1 Este conteo puede ser útil, especialmente si ha creado un dataset reutilizable, pero es posible que desee enriquecerlo con sumas. Tenga en cuenta que addmargins le permite elegir una función diferente a la suma.
t <- addmargins(table(df$pregnant,df$diabetes)) # ordenación con las sumas 6 líneas extraídas de una simple ordenación plana, núm embarazos vs estado respecto a la diabetes
---------------
neg pos Sum
0 73 38 111 ... Ediciones ENI Editorial | líder en informática
Ediciones ENI Editorial | líder en informática


